Clase 14: Introducción a pandas
El análisis y consumo de grandes cantidades de información requiere
herramientas que permitan limpiar, filtrar, agrupar los datos. Una de
las bibliotecas fundamentales para ello es pandas. pandas
contiene estructuras de datos y funciones que permiten la manipulación
de los datos en forma rápida. pandas está inspirada fuertemente en
numpy, en el sentido en que adopta en su diseño el estilo idiomático
de numpy. La diferencia principal radica en que numpy se utiliza
para conjuntos de datos homogéneos, mientras que pandas está
diseñada para trabajar con tablas o datos heterogéneos.
Otra biblioteca con características similares es polars
Input/Output con pandas
Para poder leer o escribir datos, pandas provee una serie de métodos
específicos para todos los tipos de datos estructurados usuales, ya sea
en formato texto o binario. Los métodos para leer de archivos se
reconocen por el prefijo .read_, mientras que para escribir usaremos
.to_. Para cada formato particular de archivo, estos métodos
aceptarán un conjunto de argumentos adicionales que permiten adecuar
nuestro código.
Si se usa VSCode, se puede instalar la extensión Data Wrangler que permite inspeccionar
DataFrames. Puede ser útil cuando uno trabaja con muchos datos.
JSON
JSON (JavaScript Object Notation) es un formato estándar de estructura de datos en modo texto, legible y de amplio uso en internet.
import pandas as pd
from pathlib import Path
atomos_path = Path.cwd().parent / 'data' / 'atomos'
print(atomos_path)
/Users/flavioc/Library/Mobile Documents/com~apple~CloudDocs/Documents/cursos/Python/GitLab/clase-python/data/atomos
df = pd.read_json(atomos_path / 'atomos.json')
df
| Element | Symbol | Atomic Number | Atomic Mass (u) | Density (g/cm³) | Melting Point (K) | Boiling Point (K) | Electronegativity | State at Room Temp | |
|---|---|---|---|---|---|---|---|---|---|
| 0 | Hydrogen | H | 1 | 1.0080 | 0.000090 | 14.01 | 20.28 | 2.20 | Gas |
| 1 | Helium | He | 2 | 4.0026 | 0.000179 | NaN | NaN | NaN | Gas |
| 2 | Lithium | Li | 3 | 6.9400 | 0.534000 | 453.69 | 1615.00 | 0.98 | Solid |
| 3 | Beryllium | Be | 4 | 9.0122 | 1.850000 | 1560.00 | 2742.00 | 1.57 | Solid |
| 4 | Boron | B | 5 | 10.8100 | 2.340000 | 2349.00 | 4200.00 | 2.04 | Solid |
| 5 | Carbon | C | 6 | 12.0110 | 2.267000 | 3800.00 | 4300.00 | 2.55 | Solid |
| 6 | Nitrogen | N | 7 | 14.0070 | 0.001251 | 63.15 | 77.36 | 3.04 | Gas |
| 7 | Oxygen | O | 8 | 15.9990 | 0.001429 | 54.36 | 90.20 | 3.44 | Gas |
| 8 | Fluorine | F | 9 | 18.9980 | 0.001696 | 53.48 | 85.03 | 3.98 | Gas |
| 9 | Neon | Ne | 10 | 20.1800 | 0.000900 | NaN | NaN | NaN | Gas |
df.dtypes
Element object
Symbol object
Atomic Number int64
Atomic Mass (u) float64
Density (g/cm³) float64
Melting Point (K) float64
Boiling Point (K) float64
Electronegativity float64
State at Room Temp object
dtype: object
df.columns
Index(['Element', 'Symbol', 'Atomic Number', 'Atomic Mass (u)',
'Density (g/cm³)', 'Melting Point (K)', 'Boiling Point (K)',
'Electronegativity', 'State at Room Temp'],
dtype='object')
Renombremos algunas columnas para que no tengan etiquetas tan complejas
df.rename(columns={'Density (g/cm³)': 'Density'}, inplace=True)
df.rename(columns={'Melting Point (K)': 'Melting Point'}, inplace=True)
df.columns
Index(['Element', 'Symbol', 'Atomic Number', 'Atomic Mass (u)', 'Density',
'Melting Point', 'Boiling Point (K)', 'Electronegativity',
'State at Room Temp'],
dtype='object')
Extraigamos los datos atómicos (masa y número atómico) y escribámoslo en un json:
df[['Element', 'Symbol', 'Atomic Number', 'Atomic Mass (u)']]
| Element | Symbol | Atomic Number | Atomic Mass (u) | |
|---|---|---|---|---|
| 0 | Hydrogen | H | 1 | 1.0080 |
| 1 | Helium | He | 2 | 4.0026 |
| 2 | Lithium | Li | 3 | 6.9400 |
| 3 | Beryllium | Be | 4 | 9.0122 |
| 4 | Boron | B | 5 | 10.8100 |
| 5 | Carbon | C | 6 | 12.0110 |
| 6 | Nitrogen | N | 7 | 14.0070 |
| 7 | Oxygen | O | 8 | 15.9990 |
| 8 | Fluorine | F | 9 | 18.9980 |
| 9 | Neon | Ne | 10 | 20.1800 |
df[['Element', 'Symbol', 'Atomic Number', 'Atomic Mass (u)']].to_json(atomos_path / 'prop_atomos.json')
# Guarda sólo los valores
df[['Element', 'Symbol', 'Atomic Number', 'Atomic Mass (u)']].to_json(atomos_path / 'prop_atomos_values.json', orient='values')
# Guarda los registros indexados por el índice del DataFrame
df[['Element', 'Symbol', 'Atomic Number', 'Atomic Mass (u)']].to_json(atomos_path / 'prop_atomos_index.json', orient='index')
# Guarda los registros indexados por el nombre de las columnas, valor por defecto
df[['Element', 'Symbol', 'Atomic Number', 'Atomic Mass (u)']].to_json(atomos_path / 'prop_atomos_columns.json', orient='columns')
# Guarda los registros en formato de lista de diccionarios, sin indices
df[['Element', 'Symbol', 'Atomic Number', 'Atomic Mass (u)']].to_json(atomos_path / 'prop_atomos_records.json', orient='records')
CSV
Podríamos querer escribirlos como valores separados por comas (u otro delimitador):
df[['Element', 'Symbol', 'Atomic Number', 'Atomic Mass (u)']].to_csv(atomos_path / 'prop_atomos.csv', sep='|')
Vemos que en ambos casos también guarda el índice en el archivo
Si queremos que no guarde el índice, pasamos el argumento opcional
index=False.
df.to_csv(atomos_path / 'prop_atomos_noindex.csv', sep='|', index=False)
Se puede ver que al guardar el .csv, los elementos inexistentes de
la tabla (NaN) se guardan como cadenas de caracteres vacías. Se
puede indicar otro tipo de valor para esos casos:
df.to_csv(atomos_path / 'prop_atomos_noheader2.csv', sep='|', index=False, na_rep='N/A')
Formatos binarios
En algunos casos como leer o escribir de archivos binarios, es necesario
instalar algunos módulos. Por ejemplo, si queremos leer archivos
.parquet, tenemos que instalar pyarrow.
.parquet:conda install pyarrow.xls(x):conda install openpyxl xlrdhdf5:conda install pytables
df.to_parquet(atomos_path / 'prop_atomos.parquet')
dfp = pd.read_parquet(atomos_path / 'prop_atomos.parquet')
dfp
| Element | Symbol | Atomic Number | Atomic Mass (u) | Density | Melting Point | Boiling Point (K) | Electronegativity | State at Room Temp | |
|---|---|---|---|---|---|---|---|---|---|
| 0 | Hydrogen | H | 1 | 1.0080 | 0.000090 | 14.01 | 20.28 | 2.20 | Gas |
| 1 | Helium | He | 2 | 4.0026 | 0.000179 | NaN | NaN | NaN | Gas |
| 2 | Lithium | Li | 3 | 6.9400 | 0.534000 | 453.69 | 1615.00 | 0.98 | Solid |
| 3 | Beryllium | Be | 4 | 9.0122 | 1.850000 | 1560.00 | 2742.00 | 1.57 | Solid |
| 4 | Boron | B | 5 | 10.8100 | 2.340000 | 2349.00 | 4200.00 | 2.04 | Solid |
| 5 | Carbon | C | 6 | 12.0110 | 2.267000 | 3800.00 | 4300.00 | 2.55 | Solid |
| 6 | Nitrogen | N | 7 | 14.0070 | 0.001251 | 63.15 | 77.36 | 3.04 | Gas |
| 7 | Oxygen | O | 8 | 15.9990 | 0.001429 | 54.36 | 90.20 | 3.44 | Gas |
| 8 | Fluorine | F | 9 | 18.9980 | 0.001696 | 53.48 | 85.03 | 3.98 | Gas |
| 9 | Neon | Ne | 10 | 20.1800 | 0.000900 | NaN | NaN | NaN | Gas |
Web
# URL que contiene una tabla HTML
url = "https://en.wikipedia.org/wiki/List_of_countries_by_population"
# Leer todas las tablas de la página
tablas = pd.read_html(url)
print(f"Encontré {len(tablas)} tablas")
print(type(tablas))
# La función devuelve una lista de DataFrames, cada uno correspondiente a una tabla en la página
# Normalmente querrás seleccionar una tabla específica
tabla_poblacion = tablas[0] # Primera tabla de la página
# Mostrar las primeras filas
tabla_poblacion.head()
Encontré 3 tablas
<class 'list'>
| Location | Population | % of world | Date | Source (official or from the United Nations) | Notes | |
|---|---|---|---|---|---|---|
| 0 | World | 8137000000 | 100% | 10 Apr 2025 | UN projection[1][3] | NaN |
| 1 | India | 1413324000 | 17.3% | 1 Mar 2025 | Official projection[4] | [b] |
| 2 | China | 1408280000 | 17.2% | 31 Dec 2024 | Official estimate[5] | [c] |
| 3 | United States | 340110988 | 4.2% | 1 Jul 2024 | Official estimate[6] | [d] |
| 4 | Indonesia | 282477584 | 3.5% | 30 Jun 2024 | National annual projection[7] | NaN |
Cuando se pone difícil
En muchas situaciones los archivos pueden tener datos estructurados de
alguna manera que no permiten directamente su lectura con pandas.
smn_path = Path.cwd().parent / 'data' / 'smn'
pronostico = pd.read_csv(smn_path / 'pronostico_header4.txt')
pronostico
| ************************************************************************************************ | |
|---|---|
| Producto basado en un modelo de pronóstico numérico del tiempo | |
| por lo tanto puede diferir del pronostico emitido por el SMN | NaN |
| ************************************************************************************************ | NaN |
| AEROPARQUE | NaN |
| ================================================================================================ | NaN |
| ... | ... |
| 06/DIC/2024 12Hs. 13.1 314 | 12 0.0 | NaN |
| 06/DIC/2024 15Hs. 16.2 297 | 15 0.0 | NaN |
| 06/DIC/2024 18Hs. 15.2 304 | 23 0.0 | NaN |
| 06/DIC/2024 21Hs. 9.1 308 | 17 0.0 | NaN |
| ================================================================================================ | NaN |
187 rows × 1 columns
Probemos usando read_fwf, que es capaz de leer tablas con columnas
de ancho fijo:
pronostico = pd.read_fwf(smn_path / 'pronostico_header4.txt',skiprows=5)
pronostico.head(10)
| AEROPARQUE | |
|---|---|
| 0 | ==============================================... |
| 1 | FECHA * TEMPERATURA VIENTO ... |
| 2 | (DIR | KM/H) |
| 3 | ==============================================... |
| 4 | 02/DIC/2024 00Hs. 16.0 236 | 17 ... |
| 5 | 02/DIC/2024 03Hs. 14.4 196 | 18 ... |
| 6 | 02/DIC/2024 06Hs. 10.6 190 | 8 ... |
| 7 | 02/DIC/2024 09Hs. 14.9 221 | 11 ... |
| 8 | 02/DIC/2024 12Hs. 19.2 249 | 12 ... |
| 9 | 02/DIC/2024 15Hs. 21.6 241 | 15 ... |
Como se esperaba, no parece ser la solución porque la estructura del archivo no es estrictamente de columnas de ancho fijo, sino de secciones de columnas de ancho fijo. Entonces tenemos que preprocesar el archivo para llegar a obtener esas secciones:
with open(smn_path / 'pronostico_5dias20250408.txt', 'r') as file:
content = file.read()
# Dividir el contenido en secciones por aeropuerto
sep = "================================================================================================"
sections = content.split(sep)
for i,s in enumerate(sections[0:5]):
print(f"Sección {i} {'-'*80}")
print(f"{s}")
Sección 0 --------------------------------------------------------------------------------
********************************************************************************************
Producto basado en un modelo de pronóstico numérico del tiempo,
por lo tanto puede diferir del pronostico emitido por el SMN
********************************************************************************************
AEROPARQUE
Sección 1 --------------------------------------------------------------------------------
FECHA * TEMPERATURA VIENTO PRECIPITACION(mm)
(DIR | KM/H)
Sección 2 --------------------------------------------------------------------------------
08/ABR/2025 00Hs. 15.4 116 | 27 7.5
08/ABR/2025 03Hs. 15.6 112 | 25 3.0
08/ABR/2025 06Hs. 16.0 114 | 26 3.0
08/ABR/2025 09Hs. 16.3 115 | 26 1.7
08/ABR/2025 12Hs. 16.6 123 | 22 1.1
08/ABR/2025 15Hs. 17.0 123 | 24 0.9
08/ABR/2025 18Hs. 16.7 117 | 24 1.8
08/ABR/2025 21Hs. 16.6 104 | 22 1.4
09/ABR/2025 00Hs. 16.7 100 | 22 0.6
09/ABR/2025 03Hs. 16.2 110 | 21 1.2
09/ABR/2025 06Hs. 16.0 107 | 18 1.3
09/ABR/2025 09Hs. 16.1 99 | 18 1.9
09/ABR/2025 12Hs. 17.3 99 | 14 0.7
09/ABR/2025 15Hs. 19.9 106 | 19 0.4
09/ABR/2025 18Hs. 18.2 104 | 17 0.0
09/ABR/2025 21Hs. 17.1 108 | 16 0.0
10/ABR/2025 00Hs. 17.1 109 | 15 0.0
10/ABR/2025 03Hs. 17.6 104 | 16 0.0
10/ABR/2025 06Hs. 17.0 94 | 13 0.0
10/ABR/2025 09Hs. 18.2 98 | 13 0.0
10/ABR/2025 12Hs. 19.7 102 | 11 0.1
10/ABR/2025 15Hs. 21.3 138 | 12 0.0
10/ABR/2025 18Hs. 19.5 120 | 14 0.0
10/ABR/2025 21Hs. 18.6 109 | 8 0.2
11/ABR/2025 00Hs. 17.2 22 | 4 0.0
11/ABR/2025 03Hs. 14.8 35 | 6 0.0
11/ABR/2025 06Hs. 15.2 26 | 4 0.0
11/ABR/2025 09Hs. 17.5 30 | 2 0.0
11/ABR/2025 12Hs. 19.6 6 | 3 0.2
11/ABR/2025 15Hs. 22.8 326 | 3 0.0
11/ABR/2025 18Hs. 20.1 187 | 4 0.0
11/ABR/2025 21Hs. 18.2 115 | 3 0.0
12/ABR/2025 00Hs. 16.6 292 | 3 0.0
12/ABR/2025 03Hs. 15.1 254 | 6 0.0
12/ABR/2025 06Hs. 14.4 239 | 6 0.0
12/ABR/2025 09Hs. 17.6 213 | 6 0.0
12/ABR/2025 12Hs. 22.4 149 | 7 0.0
12/ABR/2025 15Hs. 22.8 123 | 8 0.0
12/ABR/2025 18Hs. 19.5 72 | 4 0.0
12/ABR/2025 21Hs. 17.0 95 | 8 0.0
Sección 3 --------------------------------------------------------------------------------
AZUL_AERO
Sección 4 --------------------------------------------------------------------------------
FECHA * TEMPERATURA VIENTO PRECIPITACION(mm)
(DIR | KM/H)
print("Estación : ", sections[0])
print("Header : ", sections[1])
print("Pronóstico : ", sections[2])
Estación : ********************************************************************************************
Producto basado en un modelo de pronóstico numérico del tiempo,
por lo tanto puede diferir del pronostico emitido por el SMN
********************************************************************************************
AEROPARQUE
Header :
FECHA * TEMPERATURA VIENTO PRECIPITACION(mm)
(DIR | KM/H)
Pronóstico :
08/ABR/2025 00Hs. 15.4 116 | 27 7.5
08/ABR/2025 03Hs. 15.6 112 | 25 3.0
08/ABR/2025 06Hs. 16.0 114 | 26 3.0
08/ABR/2025 09Hs. 16.3 115 | 26 1.7
08/ABR/2025 12Hs. 16.6 123 | 22 1.1
08/ABR/2025 15Hs. 17.0 123 | 24 0.9
08/ABR/2025 18Hs. 16.7 117 | 24 1.8
08/ABR/2025 21Hs. 16.6 104 | 22 1.4
09/ABR/2025 00Hs. 16.7 100 | 22 0.6
09/ABR/2025 03Hs. 16.2 110 | 21 1.2
09/ABR/2025 06Hs. 16.0 107 | 18 1.3
09/ABR/2025 09Hs. 16.1 99 | 18 1.9
09/ABR/2025 12Hs. 17.3 99 | 14 0.7
09/ABR/2025 15Hs. 19.9 106 | 19 0.4
09/ABR/2025 18Hs. 18.2 104 | 17 0.0
09/ABR/2025 21Hs. 17.1 108 | 16 0.0
10/ABR/2025 00Hs. 17.1 109 | 15 0.0
10/ABR/2025 03Hs. 17.6 104 | 16 0.0
10/ABR/2025 06Hs. 17.0 94 | 13 0.0
10/ABR/2025 09Hs. 18.2 98 | 13 0.0
10/ABR/2025 12Hs. 19.7 102 | 11 0.1
10/ABR/2025 15Hs. 21.3 138 | 12 0.0
10/ABR/2025 18Hs. 19.5 120 | 14 0.0
10/ABR/2025 21Hs. 18.6 109 | 8 0.2
11/ABR/2025 00Hs. 17.2 22 | 4 0.0
11/ABR/2025 03Hs. 14.8 35 | 6 0.0
11/ABR/2025 06Hs. 15.2 26 | 4 0.0
11/ABR/2025 09Hs. 17.5 30 | 2 0.0
11/ABR/2025 12Hs. 19.6 6 | 3 0.2
11/ABR/2025 15Hs. 22.8 326 | 3 0.0
11/ABR/2025 18Hs. 20.1 187 | 4 0.0
11/ABR/2025 21Hs. 18.2 115 | 3 0.0
12/ABR/2025 00Hs. 16.6 292 | 3 0.0
12/ABR/2025 03Hs. 15.1 254 | 6 0.0
12/ABR/2025 06Hs. 14.4 239 | 6 0.0
12/ABR/2025 09Hs. 17.6 213 | 6 0.0
12/ABR/2025 12Hs. 22.4 149 | 7 0.0
12/ABR/2025 15Hs. 22.8 123 | 8 0.0
12/ABR/2025 18Hs. 19.5 72 | 4 0.0
12/ABR/2025 21Hs. 17.0 95 | 8 0.0
La lista sections contiene secciones que corresponden a una estación
meteorológica, cuyos nombres son:
print(sections[0].split('\n')[-2].strip()) # tratamiento especial para remover el comentario
print(sections[3].strip())
print(sections[6].strip())
AEROPARQUE
AZUL_AERO
BAHIA_BLANCA_AERO
La primer sección debe trabajarse a mano debido al comentario que
posee el archivo
Las siguientes serían las secciones de headers que en principio no nos harían falta:
print(sections[1].strip())
print(sections[4].strip())
print(sections[7].strip())
FECHA * TEMPERATURA VIENTO PRECIPITACION(mm)
(DIR | KM/H)
FECHA * TEMPERATURA VIENTO PRECIPITACION(mm)
(DIR | KM/H)
FECHA * TEMPERATURA VIENTO PRECIPITACION(mm)
(DIR | KM/H)
Finalmente tenemos las secciones con los datos:
print(sections[2].split('\n')[:5])
print(sections[5].split('\n')[:5])
print(sections[8].split('\n')[:5])
['', ' 08/ABR/2025 00Hs. 15.4 116 | 27 7.5 ', ' 08/ABR/2025 03Hs. 15.6 112 | 25 3.0 ', ' 08/ABR/2025 06Hs. 16.0 114 | 26 3.0 ', ' 08/ABR/2025 09Hs. 16.3 115 | 26 1.7 ']
['', ' 08/ABR/2025 00Hs. 10.3 109 | 15 0.0 ', ' 08/ABR/2025 03Hs. 9.3 124 | 12 0.0 ', ' 08/ABR/2025 06Hs. 8.9 134 | 11 0.0 ', ' 08/ABR/2025 09Hs. 12.6 122 | 18 0.0 ']
['', ' 08/ABR/2025 00Hs. 13.5 35 | 20 0.0 ', ' 08/ABR/2025 03Hs. 11.7 31 | 16 0.0 ', ' 08/ABR/2025 06Hs. 10.7 42 | 12 0.0 ', ' 08/ABR/2025 09Hs. 12.6 50 | 15 0.0 ']
Ahora sí podemos usar read_fwf para transformar las secciones en
DataFrames. Para ello tenemos que convertir cada section (que
es un string) en un tipo de buffer en memoria que se comporte como un
archivo. Obsérvese que si hacemos:
from io import StringIO
with StringIO(sections[5]) as data_io:
df_malo = pd.read_fwf(data_io)
df_malo.head()
| 08/ABR/2025 | 00Hs. | 10.3 | 109 | | | 15 | 0.0 | |
|---|---|---|---|---|---|---|---|
| 0 | 08/ABR/2025 | 03Hs. | 9.3 | 124 | | | 12 | 0.0 |
| 1 | 08/ABR/2025 | 06Hs. | 8.9 | 134 | | | 11 | 0.0 |
| 2 | 08/ABR/2025 | 09Hs. | 12.6 | 122 | | | 18 | 0.0 |
| 3 | 08/ABR/2025 | 12Hs. | 19.4 | 95 | | | 22 | 0.0 |
| 4 | 08/ABR/2025 | 15Hs. | 21.4 | 99 | | | 28 | 0.0 |
Es decir que la primer fila del stream es tomada como los nombres de
las columnas. Tenemos que pasar el argumento opcional names para
definir los nombres de las columnas:
with StringIO(sections[2]) as data_io:
df = pd.read_fwf(data_io,names=['fecha','h','t','v_dir','l','v_vel','precip'])
df.head()
| fecha | h | t | v_dir | l | v_vel | precip | |
|---|---|---|---|---|---|---|---|
| 0 | 08/ABR/2025 | 00Hs. | 15.4 | 116 | | | 27 | 7.5 |
| 1 | 08/ABR/2025 | 03Hs. | 15.6 | 112 | | | 25 | 3.0 |
| 2 | 08/ABR/2025 | 06Hs. | 16.0 | 114 | | | 26 | 3.0 |
| 3 | 08/ABR/2025 | 09Hs. | 16.3 | 115 | | | 26 | 1.7 |
| 4 | 08/ABR/2025 | 12Hs. | 16.6 | 123 | | | 22 | 1.1 |
with StringIO(sections[2]) as data_io:
df_auto = pd.read_fwf(data_io,header=None)
df_auto.head()
| 0 | 1 | 2 | 3 | 4 | 5 | 6 | |
|---|---|---|---|---|---|---|---|
| 0 | 08/ABR/2025 | 00Hs. | 15.4 | 116 | | | 27 | 7.5 |
| 1 | 08/ABR/2025 | 03Hs. | 15.6 | 112 | | | 25 | 3.0 |
| 2 | 08/ABR/2025 | 06Hs. | 16.0 | 114 | | | 26 | 3.0 |
| 3 | 08/ABR/2025 | 09Hs. | 16.3 | 115 | | | 26 | 1.7 |
| 4 | 08/ABR/2025 | 12Hs. | 16.6 | 123 | | | 22 | 1.1 |
Para terminar podemos eliminar la columna l que no aporta
información:
df.drop(columns=['l'],inplace=True)
df.head()
| fecha | h | t | v_dir | v_vel | precip | |
|---|---|---|---|---|---|---|
| 0 | 08/ABR/2025 | 00Hs. | 15.4 | 116 | 27 | 7.5 |
| 1 | 08/ABR/2025 | 03Hs. | 15.6 | 112 | 25 | 3.0 |
| 2 | 08/ABR/2025 | 06Hs. | 16.0 | 114 | 26 | 3.0 |
| 3 | 08/ABR/2025 | 09Hs. | 16.3 | 115 | 26 | 1.7 |
| 4 | 08/ABR/2025 | 12Hs. | 16.6 | 123 | 22 | 1.1 |
Ejercicios 14 (a)
Complete la tabla de datos del ejemplo de clase. Para ello utilice el archivo con los pronósticos completos para todas las estaciones,
pronostico_5dias20250408.txty
Agregue una columna con el nombre de la estación al DataFrame
Procese todas las estaciones
Estructuras de datos en pandas: Series
Las series son un objeto unidimensional que contiene una secuencia de valores del mismo tipo y un conjunto de etiquetas denominado índice:
import numpy as np
import pandas as pd
s = pd.Series([1,4,4,2])
print(s)
print(type(s))
print(s.dtype)
0 1
1 4
2 4
3 2
dtype: int64
<class 'pandas.core.series.Series'>
int64
En este ejemplo, s es una serie que contiene elementos int64.
Los tipos de datos son extensiones de aquellos que utiliza
numpy.
Notar que usando directamente la función
Se puede acceder a los valores de la serie con el método .array, y a
los índices con el método .index:
s.array
<NumpyExtensionArray>
[1, 4, 4, 2]
Length: 4, dtype: int64
El tipo NumpyExtensionArray es una clase que encapsula al tipo array
de numpy, pero ha permitido agregarle flexibilidad para acomodar las
características de datos más generales.
Por otra parte, los índices se obtienen como:
s.index
RangeIndex(start=0, stop=4, step=1)
Los índices son inmutables, en el mismo sentido que los caracteres de un string. Es decir, no se pueden cambiar ciertos índices, pero se puede reasignar el índice de una serie completamente:
print(s.index[2])
s.index[2] = 5
2
---------------------------------------------------------------------------
TypeError Traceback (most recent call last)
Cell In[5], line 2
1 print(s.index[2])
----> 2 s.index[2] = 5
File /usr/lib64/python3.13/site-packages/pandas/core/indexes/base.py:5371, in Index.__setitem__(self, key, value)
5369 @final
5370 def __setitem__(self, key, value) -> None:
-> 5371 raise TypeError("Index does not support mutable operations")
TypeError: Index does not support mutable operations
nuevo_index = index=['a','b','c','d']
s.index = nuevo_index
print(s)
a 1
b 4
c 4
d 2
dtype: int64
Se puede también construir una nueva serie indicando los índices
específicamente en el constructor con el argumento index=:
s2 = pd.Series([1,4,4,2], index=['arquero','defensores','medios','delanteros'])
print(s2)
arquero 1
defensores 4
medios 4
delanteros 2
dtype: int64
Existen otras maneras de crear series en pandas, como por ejemplo a
partir de arreglos de numpy
rnd = pd.Series(np.random.randn(5))
print(rnd)
0 0.370947
1 1.600918
2 0.161127
3 -0.929294
4 -0.121632
dtype: float64
A partir de otras variables
values = [1,2,3,4,5]
index = ['a','b','c','d','e']
s3 = pd.Series(values, index=index)
s3
a 1
b 2
c 3
d 4
e 5
dtype: int64
o usando diccionarios:
d = { 1: "a", 2: "b", 3: "c", 4: "d", 5: "e" }
s4 = pd.Series(d,dtype='string')
print(s4)
1 a
2 b
3 c
4 d
5 e
dtype: string
Asimismo, se puede usar el argumento dtype (igual que en NumPy) para
indicar el tipo de dato que se quiere utilizar al crear la serie. Pandas
soporta los tipos de datos de NumPy, además de proveer sus propios tipos
de datos (por ejemplo, string) propios
n = pd.Series([4,5,6], dtype='string')
print(n)
print(n[0]+n[1])
0 4
1 5
2 6
dtype: string
45
Accediendo a los valores
Se puede acceder a los valores de una serie a través del índice como si fuera un array:
s = pd.Series([ 4, 8, 15, 16, 23, 42 ],index = ['a','b','c','d','e','f'])
print(s)
a 4
b 8
c 15
d 16
e 23
f 42
dtype: int64
print(f"Tercer elemento: {s.array[2]}")
s.array[2] = 108
print(s.array)
print(f"Con un rango:\n{s[1:3]}")
print(f"Con los índices:\n{s[['a','c']]}")
print(f"Con una máscara:\n{s[s > 100]}")
Tercer elemento: 15
<NumpyExtensionArray>
[4, 8, 108, 16, 23, 42]
Length: 6, dtype: int64
Con un rango:
b 8
c 108
dtype: int64
Con los índices:
a 4
c 108
dtype: int64
Con una máscara:
c 108
dtype: int64
print(f"Con una lista de índices:\n{s[[0,1]]}")
Con una lista de índices:
a 4
b 8
dtype: int64
/tmp/ipykernel_213536/1722261588.py:1: FutureWarning: Series.__getitem__ treating keys as positions is deprecated. In a future version, integer keys will always be treated as labels (consistent with DataFrame behavior). To access a value by position, use ser.iloc[pos]
print(f"Con una lista de índices:n{s[[0,1]]}")
print(f"Con una lista de índices:\n{s.iloc[[0,3]]}")
print(f"Con una lista de índices:\n{s[ s.index[[0,3]] ]}")
Con una lista de índices:
a 4
d 16
dtype: int64
Con una lista de índices:
a 4
d 16
dtype: int64
Modificando valores
Para modificar un valor dentro de una serie de pandas, se accede al
mismo con la etiqueta correspondiente:
v = pd.Series([1,2,3,4,5], index=['a','b','c','d','e'])
print(v)
v['b'] = 100
print(v)
a 1
b 2
c 3
d 4
e 5
dtype: int64
a 1
b 100
c 3
d 4
e 5
dtype: int64
En caso en que dicha etiqueta no exista dentro del índice de la serie, se agrega el valor:
v['f'] = 200
print(v)
a 1
b 100
c 3
d 4
e 5
f 200
dtype: int64
Para eliminar valores, se utiliza el método .drop. Por defecto se
genera una nueva serie con el valor eliminado, para poder eliminar el
valor en la misma serie, es necesario usar el argumento opcional
inplace como True.
w = v.drop('b')
print(v)
print(w)
a 1
b 100
c 3
d 4
e 5
f 200
dtype: int64
a 1
c 3
d 4
e 5
f 200
dtype: int64
v.drop('b', inplace=True)
print(v)
a 1
c 3
d 4
e 5
f 200
dtype: int64
# Generando un nuevo objeto
print(v==w)
# Comparando objetos
print(f"v is w: {v is w}")
print(f"v equals w: {v.equals(w)}")
a True
c True
d True
e True
f True
dtype: bool
v is w: False
v equals w: True
v.drop(['b'], inplace=True) # KeyError: "['b'] not found in axis"
---------------------------------------------------------------------------
KeyError Traceback (most recent call last)
Cell In[21], line 1
----> 1 v.drop(['b'], inplace=True) # KeyError: "['b'] not found in axis"
File /usr/lib64/python3.13/site-packages/pandas/core/series.py:5347, in Series.drop(self, labels, axis, index, columns, level, inplace, errors)
5250 def drop(
5251 self,
5252 labels: IndexLabel | None = None,
(...)
5259 errors: IgnoreRaise = "raise",
5260 ) -> Series | None:
5261 """
5262 Return Series with specified index labels removed.
5263
(...)
5345 dtype: float64
5346 """
-> 5347 return super().drop(
5348 labels=labels,
5349 axis=axis,
5350 index=index,
5351 columns=columns,
5352 level=level,
5353 inplace=inplace,
5354 errors=errors,
5355 )
File /usr/lib64/python3.13/site-packages/pandas/core/generic.py:4785, in NDFrame.drop(self, labels, axis, index, columns, level, inplace, errors)
4783 for axis, labels in axes.items():
4784 if labels is not None:
-> 4785 obj = obj._drop_axis(labels, axis, level=level, errors=errors)
4787 if inplace:
4788 self._update_inplace(obj)
File /usr/lib64/python3.13/site-packages/pandas/core/generic.py:4827, in NDFrame._drop_axis(self, labels, axis, level, errors, only_slice)
4825 new_axis = axis.drop(labels, level=level, errors=errors)
4826 else:
-> 4827 new_axis = axis.drop(labels, errors=errors)
4828 indexer = axis.get_indexer(new_axis)
4830 # Case for non-unique axis
4831 else:
File /usr/lib64/python3.13/site-packages/pandas/core/indexes/base.py:7070, in Index.drop(self, labels, errors)
7068 if mask.any():
7069 if errors != "ignore":
-> 7070 raise KeyError(f"{labels[mask].tolist()} not found in axis")
7071 indexer = indexer[~mask]
7072 return self.delete(indexer)
KeyError: "['b'] not found in axis"
Tipos de datos
Como último comentario, obsérvese que si uno pretende crear una serie
con valores de distinto tipo, el tipo de la serie se promueve al tipo
general object, sin embargo cada elemento es reconocido con su tipo
particular:
s = pd.Series([1,'a','b','c'])
print(s)
for index,elem in s.items():
print(f"Tipo de objeto del elemento {index}: {type(s[index])}")
0 1
1 a
2 b
3 c
dtype: object
Tipo de objeto del elemento 0: <class 'int'>
Tipo de objeto del elemento 1: <class 'str'>
Tipo de objeto del elemento 2: <class 'str'>
Tipo de objeto del elemento 3: <class 'str'>
print(f"Suma de enteros: {s[0] + 3}")
print(f"Suma de enteros: {s[1] + 3}") # Error
Suma de enteros: 4
---------------------------------------------------------------------------
TypeError Traceback (most recent call last)
Cell In[23], line 2
1 print(f"Suma de enteros: {s[0] + 3}")
----> 2 print(f"Suma de enteros: {s[1] + 3}") # Error
TypeError: can only concatenate str (not "int") to str
print(f"Concat de str : {s[1] + 'd'}")
print(f"Concat de str : {s[1] + 3}") # Error
Concat de str : ad
---------------------------------------------------------------------------
TypeError Traceback (most recent call last)
Cell In[24], line 2
1 print(f"Concat de str : {s[1] + 'd'}")
----> 2 print(f"Concat de str : {s[1] + 3}") # Error
TypeError: can only concatenate str (not "int") to str
Si bien entonces se pueden usar series con objetos con distintos tipos, se recomienda que el tipo de dato sea homogéneo.
Valores que faltan
Es muy común al procesar datos que uno encuentre valores que no existen.
La manera en que pandas representa estos datos faltantes es a través
de np.nan, el tipo de dato de numpy que representa not a
number, para aquellos tipos de datos heredados de numpy:
snan = pd.Series([1,4,np.nan,2])
print(snan)
0 1.0 1 4.0 2 NaN 3 2.0 dtype: float64 Notar que si bien los datos existentes son de tipo entero, al utilizarnp.nanpara representar un dato inexistente, el tipo de dato de la serie se promueve afloat64.
De la misma forma, se puede usar None para representar el dato
faltante:
snone = pd.Series([1,4,None,5])
print(snone)
0 1.0
1 4.0
2 NaN
3 5.0
dtype: float64
En el caso de cadenas de caracteres, se puede usar np.nan o
None. Si no se indica el tipo de dato a través de dtype, se
promueve el tipo de dato de la serie a object, como sucede por
defecto.
smixed_str = pd.Series(['a','b',np.nan,None,'d'])
smixed_str
0 a
1 b
2 NaN
3 None
4 d
dtype: object
Esto tiene el inconveniente evidente de que el elemento faltante no está
representado unívocamente por un solo valor. Para solventar este
problema, se puede crear la serie con el tipo string:
smixed_str2 = pd.Series(['a','b',np.nan,None,'d'],dtype='string')
print(smixed_str2)
0 a
1 b
2 <NA>
3 <NA>
4 d
dtype: string
En este caso, el singlete NA representa unívocamente el dato
faltante. En cualquier caso, se cuenta con el método .isna, que
retorna una serie de boolean donde los valores faltantes son
verdaderos (True):
print(smixed_str.isna())
print(smixed_str2.isna())
0 False
1 False
2 True
3 True
4 False
dtype: bool
0 False
1 False
2 True
3 True
4 False
dtype: bool
Atención, la cadena de caracteres vacía '' NO se considera un
dato inexistente.
Puede ser útil en algunos casos poder filtrar los datos inexistentes.
Para eso se utiliza el método .dropna, que crea una nueva serie sin
dichos datos:
print(smixed_str)
print(smixed_str.dropna())
0 a
1 b
2 NaN
3 None
4 d
dtype: object
0 a
1 b
4 d
dtype: object
Operando con series
Las operaciones con series han sido diseñadas para que sean compatibles con NumPy, y sigan las convenciones de Python. He aquí algunos ejemplos:
ones = pd.Series(np.ones(8))*0.5
print(ones)
t = pd.Series(np.random.rand(8))
print(t)
print(t.mean())
0 0.5
1 0.5
2 0.5
3 0.5
4 0.5
5 0.5
6 0.5
7 0.5
dtype: float64
0 0.855919
1 0.316680
2 0.188950
3 0.535831
4 0.767803
5 0.836171
6 0.225345
7 0.559054
dtype: float64
0.5357193052760575
shifted = t - ones
print(shifted)
print(shifted.mean())
print(np.abs(shifted))
0 0.355919
1 -0.183320
2 -0.311050
3 0.035831
4 0.267803
5 0.336171
6 -0.274655
7 0.059054
dtype: float64
0.0357193052760575
0 0.355919
1 0.183320
2 0.311050
3 0.035831
4 0.267803
5 0.336171
6 0.274655
7 0.059054
dtype: float64
Además de poder interactuar con NumPy en forma transparente, existen algunos métodos útiles para trabajar con los datos de una serie:
data = {'Pedro':19, 'Oscar': 30, 'Carlos': 27, 'David': 26}
seru = pd.Series(data)
print("Person Series:\n")
print(seru)
# Perform operations on the Series
print("Edad promedio:", seru.mean())
print("Edad del más viejo :", seru.max())
print("El más viejo :", seru.idxmax())
Person Series:
Pedro 19
Oscar 30
Carlos 27
David 26
dtype: int64
Edad promedio: 25.5
Edad del más viejo : 30
El más viejo : Oscar
temperaturas_ciudades = {
"Buenos Aires": 17.6,
"Córdoba": 18.0,
"Rosario": 18.0,
"Mendoza": 16.0,
"Santa Fe": 21.3
}
temp = pd.Series(temperaturas_ciudades)
print(temp)
Buenos Aires 17.6
Córdoba 18.0
Rosario 18.0
Mendoza 16.0
Santa Fe 21.3
dtype: float64
otras_ciudades = ['Buenos Aires', 'Córdoba', 'Rosario', 'Mendoza', 'Salta']
temp = pd.Series(temperaturas_ciudades, index=otras_ciudades)
print(temp)
Buenos Aires 17.6
Córdoba 18.0
Rosario 18.0
Mendoza 16.0
Salta NaN
dtype: float64
temp_ciudades2 = {
"Mendoza": 20.0,
"Salta": 17.2,
"Santa Fe": 18.6,
"San Juan": 19.3
}
temp2 = pd.Series(temp_ciudades2)
temp2
Mendoza 20.0
Salta 17.2
Santa Fe 18.6
San Juan 19.3
dtype: float64
print(temp)
Buenos Aires 17.6
Córdoba 18.0
Rosario 18.0
Mendoza 16.0
Salta NaN
dtype: float64
print((temp + temp2)/2)
Buenos Aires NaN
Córdoba NaN
Mendoza 18.0
Rosario NaN
Salta NaN
San Juan NaN
Santa Fe NaN
dtype: float64
Misceláneas
Algunas otras operaciones interesantes:
'Rosario' in temp2
False
temp2_dict = temp2.to_dict()
print(temp2_dict)
print(type(temp2_dict))
{'Mendoza': 20.0, 'Salta': 17.2, 'Santa Fe': 18.6, 'San Juan': 19.3}
<class 'dict'>
temp2_json = temp2.to_json()
print(temp2_json)
print(type(temp2_json))
{"Mendoza":20.0,"Salta":17.2,"Santa Fe":18.6,"San Juan":19.3}
<class 'str'>
Ejercicios 14(b)
Si no lo ha adivinado, los datos de la serie
serucorresponden a las edades de los integrantes de la banda Serú Girán, al momento de conformarse, en el año 1978. Encuentre los años de nacimiento de cada uno de sus integrantes.Una de las funciones más usadas en redes neuronales es softmax, cuyo objetivo es convertir los resultados de las distintas etapas del procesamiento de una red en probabilidades. Los primeros pueden ser números reales de cualquier valor, mientras que las probabilidades deben estar acotadas al intervalo [0,1]. La función softmax aplicada a un conjunto de valores \(z_1,\cdots,z_n\) se calcula como
\[\sigma(z_i) = \frac {e^{z_i}} { \sum_{i=1}^n e^{z_i}}\]Cree una serie con 10 valores al azar entre 0 y 5
Obtenga la serie resultante de la aplicación de softmax a la serie anterior
El siguiente es un diccionario que representa el consumo eléctrico mensual de ciertos artefactos eléctricos:
consumo_electrico = { "Artefacto": [ "Heladera", "Lavarropa", "Microondas", "Aire Acondicionado", "Televisor", "Computadora", "Lámpara LED", "Secador de Pelo", "Horno Eléctrico", "Ventilador" ], "Consumo Promedio (kWh/mes)": [ 30, 10, 15, 120, 20, 12, 2, 3, 25, 8 ] }
Obtenga los tres artefactos de mayor consumo.
Encuentre los artefactos que consumen más de 15 kWH/mes.
Calcule el consumo anual de cada artefacto, y el costo que implica suponiendo que el precio del kWH/mes es de $3145.
Un problema de índices enteros. Supongamos que tenemos la serie
s = pd.Series(np.arange(3.0))
es decir,
0 0.0 1 1.0 2 2.0 dtype: float64
¿Cuál es el resultado esperado de s[-1]? ¿Cuál es el real? Evalúe
s[-2:-1]y tambiéns.iloc[-1]. Considere ahora la series = pd.Series(np.arange(3.0), index=['a','b','c'])
y evalúe nuevamente
s[-1]. ¿Qué conclusiones puede sacar?
Estructuras de datos en pandas: DataFrames
Hemos visto que una serie en pandas es representa una colección de
valores indexados por sendas etiquetas. La extensión natural (?) de una
serie es una colección con múltiples valores arreglados en columnas
indexados por una misma colección de etiquetas. Es decir, una tabla… La
estructura que representa tablas en pandas se llama DataFrame. Un
DataFrame tiene un índice de filas (como las series) y un índice de
columnas.
Se puede pensar un
DataFramecomo un diccionario de series que comparten un mismo índice.
Como las series, es habitual construir un DataFrame a partir de un
diccionario. Veamos el siguiente diccionario para comenzar a trabajar.
Como siempre, importamos los módulos indispensables:
import numpy as np
import pandas as pd
data = {
"Titulo": [
"La piedra filosofal",
"La cámara secreta",
"El prisionero de Azkaban",
"El cáliz de fuego",
"La orden del Fénix",
"El misterio del príncipe",
"Las reliquias de la muerte"
],
"Año de edición": [1997, 1998, 1999, 2000, 2003, 2005, 2007],
"Páginas": [223, 251, 317, 636, 766, 607, 607],
}
df = pd.DataFrame(data)
df
| Titulo | Año de edición | Páginas | |
|---|---|---|---|
| 0 | La piedra filosofal | 1997 | 223 |
| 1 | La cámara secreta | 1998 | 251 |
| 2 | El prisionero de Azkaban | 1999 | 317 |
| 3 | El cáliz de fuego | 2000 | 636 |
| 4 | La orden del Fénix | 2003 | 766 |
| 5 | El misterio del príncipe | 2005 | 607 |
| 6 | Las reliquias de la muerte | 2007 | 607 |
El diccionario de origen asigna a cada clave una lista de elementos.
Hemos generado un DataFrame a partir de un diccionario: los índices
de filas se generan automáticamente, mientras que los índices (o
etiquetas) de las columnas corresponden a las claves de cada una de las
listas del diccionario.
Si las listas que componen el diccionario de origen no son iguales,
pd.DataFramedará un errorValueError: All arrays must be of the same length.
Una conveniencia interesante en Jupyter Notebooks es que se puede imprimir la tabla en forma elegante:
print(df)
display(df)
df # también funciona
Titulo Año de edición Páginas
0 La piedra filosofal 1997 223
1 La cámara secreta 1998 251
2 El prisionero de Azkaban 1999 317
3 El cáliz de fuego 2000 636
4 La orden del Fénix 2003 766
5 El misterio del príncipe 2005 607
6 Las reliquias de la muerte 2007 607
| Titulo | Año de edición | Páginas | |
|---|---|---|---|
| 0 | La piedra filosofal | 1997 | 223 |
| 1 | La cámara secreta | 1998 | 251 |
| 2 | El prisionero de Azkaban | 1999 | 317 |
| 3 | El cáliz de fuego | 2000 | 636 |
| 4 | La orden del Fénix | 2003 | 766 |
| 5 | El misterio del príncipe | 2005 | 607 |
| 6 | Las reliquias de la muerte | 2007 | 607 |
| Titulo | Año de edición | Páginas | |
|---|---|---|---|
| 0 | La piedra filosofal | 1997 | 223 |
| 1 | La cámara secreta | 1998 | 251 |
| 2 | El prisionero de Azkaban | 1999 | 317 |
| 3 | El cáliz de fuego | 2000 | 636 |
| 4 | La orden del Fénix | 2003 | 766 |
| 5 | El misterio del príncipe | 2005 | 607 |
| 6 | Las reliquias de la muerte | 2007 | 607 |
Eventualmente uno podría querer ver sólo algunas filas:
display(df.head()) # muestra las primeras 5 filas
display(df.tail()) # muestra las últimas 5 filas
| Titulo | Año de edición | Páginas | |
|---|---|---|---|
| 0 | La piedra filosofal | 1997 | 223 |
| 1 | La cámara secreta | 1998 | 251 |
| 2 | El prisionero de Azkaban | 1999 | 317 |
| 3 | El cáliz de fuego | 2000 | 636 |
| 4 | La orden del Fénix | 2003 | 766 |
| Titulo | Año de edición | Páginas | |
|---|---|---|---|
| 2 | El prisionero de Azkaban | 1999 | 317 |
| 3 | El cáliz de fuego | 2000 | 636 |
| 4 | La orden del Fénix | 2003 | 766 |
| 5 | El misterio del príncipe | 2005 | 607 |
| 6 | Las reliquias de la muerte | 2007 | 607 |
El DataFrame usa la variable index como en las series, y la
variable columns para describir las etiquetas de las columnas. Por
ejemplo:
df2 = pd.DataFrame(data, index = ["a", "b", "c", "d", "e", "f", "g"], columns = ["Titulo", "Páginas", "Año de edición"])
df2
| Titulo | Páginas | Año de edición | |
|---|---|---|---|
| a | La piedra filosofal | 223 | 1997 |
| b | La cámara secreta | 251 | 1998 |
| c | El prisionero de Azkaban | 317 | 1999 |
| d | El cáliz de fuego | 636 | 2000 |
| e | La orden del Fénix | 766 | 2003 |
| f | El misterio del príncipe | 607 | 2005 |
| g | Las reliquias de la muerte | 607 | 2007 |
Notar que el orden de la lista de etiquetas que se pasa a columns se
mantiene al crear el DataFrame. La lista que se usa en la creación
de un DataFrame usando columns debe contener exactamente los
mismos valores que las claves del diccionario, sino generará una columna
con datos faltantes.
Para acceder a las etiquetas de filas y columnas, usamos los métodos
.index y .columns.
print(f"Indices de filas: {df2.index}")
print(f"Indices de columnas: {df2.columns}")
Indices de filas: Index(['a', 'b', 'c', 'd', 'e', 'f', 'g'], dtype='object')
Indices de columnas: Index(['Titulo', 'Páginas', 'Año de edición'], dtype='object')
Accediendo a los valores
Para acceder a las columnas podemos usar:
print(f"Valores de las filas: {df2.values}")
print(f"Valores de las columnas: {type(df2.values)}")
Valores de las filas: [['La piedra filosofal' 223 1997]
['La cámara secreta' 251 1998]
['El prisionero de Azkaban' 317 1999]
['El cáliz de fuego' 636 2000]
['La orden del Fénix' 766 2003]
['El misterio del príncipe' 607 2005]
['Las reliquias de la muerte' 607 2007]]
Valores de las columnas: <class 'numpy.ndarray'>
El resultado de esta operación nos devuelve un arreglo 2D de NumPy, cuyo
tipo de dato será el mínimo que pueda contener los tipos de datos de las
columnas que lo componen. Por ejemplo, si nuestras columnas tienen datos
en float32 y float64, el tipo del arreglo de NumPy resultante
será float64. En el caso anterior, los tipos de datos de las
columnas son:
df2.dtypes
Titulo object
Páginas int64
Año de edición int64
dtype: object
Es decir que los datos en int64 de las columnas Páginas y
Año de edición se promueven a object.
El acceso a ciertas columnas en particular se hace como en los diccionarios:
titulos = df2["Titulo"]
print("La columna 'Titulo'")
print(f"{titulos}")
print(f"Tipo de la columna 'Titulo': {type(titulos)}")
print(f"Valores de la columna 'Titulo': {titulos.values}")
print(f"Tipo de la columna 'Titulo' .values: {type(titulos.values)}")
La columna 'Titulo'
a La piedra filosofal
b La cámara secreta
c El prisionero de Azkaban
d El cáliz de fuego
e La orden del Fénix
f El misterio del príncipe
g Las reliquias de la muerte
Name: Titulo, dtype: object
Tipo de la columna 'Titulo': <class 'pandas.core.series.Series'>
Valores de la columna 'Titulo': ['La piedra filosofal' 'La cámara secreta' 'El prisionero de Azkaban'
'El cáliz de fuego' 'La orden del Fénix' 'El misterio del príncipe'
'Las reliquias de la muerte']
Tipo de la columna 'Titulo' .values: <class 'numpy.ndarray'>
Vemos que una columna en un DataFrame de pandas es representada
efectivamente por una serie, de este modo, sus valores (obtenidos
mediante .values) serán un arreglo de NumPy, como vimos antes.
En el caso en que la etiqueta de una columna no contenga espacios, se
puede acceder a la misma con el operador .:
df2.Titulo # es lo mismo que df2['Titulo']
a La piedra filosofal
b La cámara secreta
c El prisionero de Azkaban
d El cáliz de fuego
e La orden del Fénix
f El misterio del príncipe
g Las reliquias de la muerte
Name: Titulo, dtype: object
Si uno quisiera usar esta propiedad en forma exhaustiva para todas
las columnas, debería reemplazar las etiquetas de las mismas por
nombres de variables válidos en Python
df2.columns = ["titulo", "paginas", "primer_edicion"]
df2
| titulo | paginas | primer_edicion | |
|---|---|---|---|
| a | La piedra filosofal | 223 | 1997 |
| b | La cámara secreta | 251 | 1998 |
| c | El prisionero de Azkaban | 317 | 1999 |
| d | El cáliz de fuego | 636 | 2000 |
| e | La orden del Fénix | 766 | 2003 |
| f | El misterio del príncipe | 607 | 2005 |
| g | Las reliquias de la muerte | 607 | 2007 |
df2.primer_edicion
a 1997 b 1998 c 1999 d 2000 e 2003 f 2005 g 2007 Name: primer_edicion, dtype: int64 En el caso en que uno cree unDataFramedesde una base de datos, una buena práctica es mantener la consistencia entre las etiquetas de las columnas delDataFramey las de la base de datos.
Se puede acceder a una determinada fila usando las etiquetas través de
.loc o con índices enteros con .iloc:
print(f"Primer libro:\n{df2.iloc[0]}")
print(f"Segundo libro:\n{df2.loc['b']}")
print(f"Último libro:\n{df2.iloc[-1]}")
Primer libro:
titulo La piedra filosofal
paginas 223
primer_edicion 1997
Name: a, dtype: object
Segundo libro:
titulo La cámara secreta
paginas 251
primer_edicion 1998
Name: b, dtype: object
Último libro:
titulo Las reliquias de la muerte
paginas 607
primer_edicion 2007
Name: g, dtype: object
Y se puede acceder a un elemento particular de la tabla:
print(f"Título primer libro: {df2.iloc[0].titulo}")
print(f"Año de edición del segundo libro: {df2.loc['b', 'primer_edicion']}")
print(f"Año de edición del último libro: {df2.iloc[-1, 2]}")
Título primer libro: La piedra filosofal
Año de edición del segundo libro: 1998
Año de edición del último libro: 2007
La figura presenta los distintos tipos de acceso a los datos de un
DataFrame
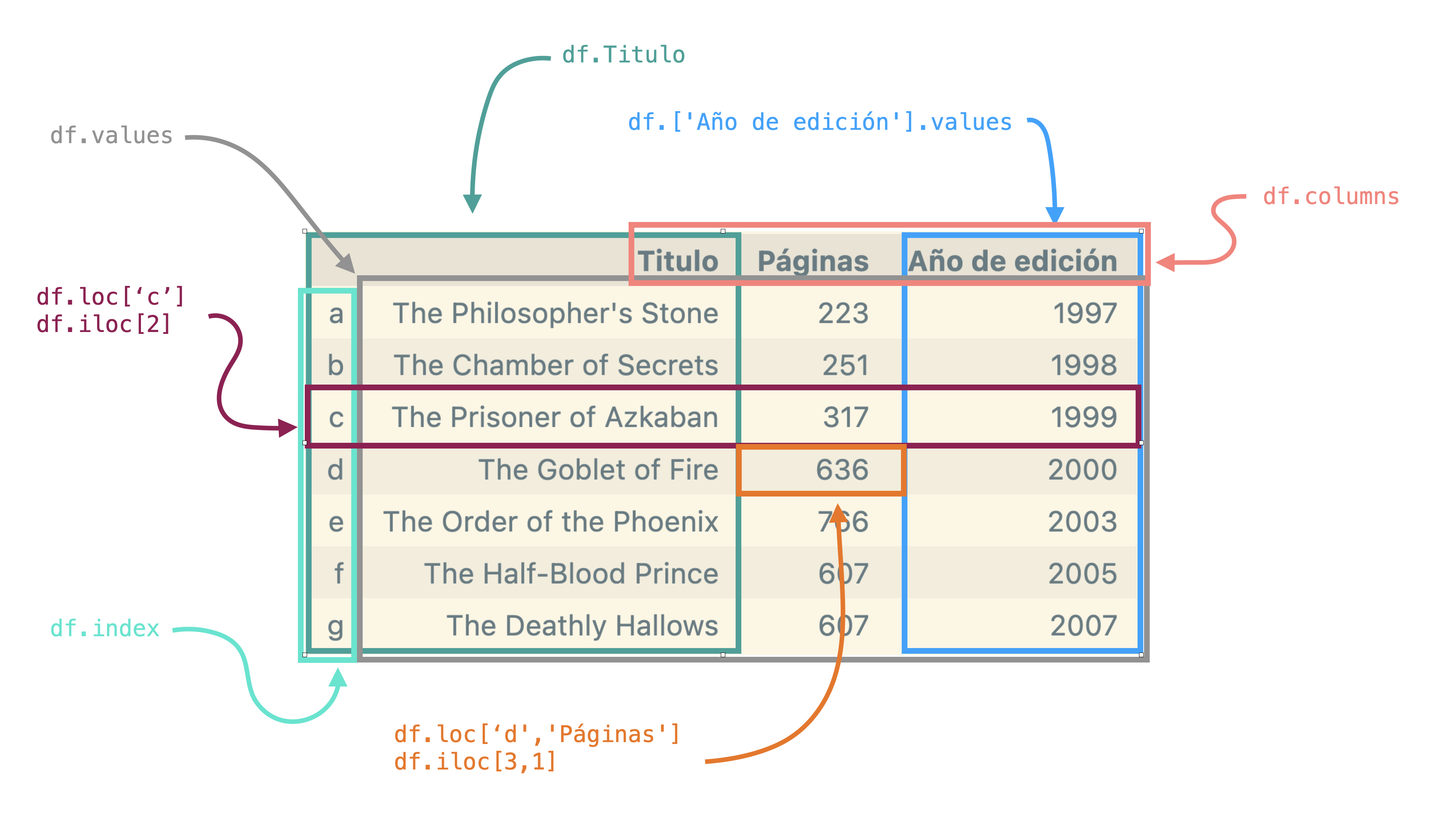
Nota
Si se quisiera acceder solo a ciertas regiones de la tabla, se pueden reemplazar los valores individuales por rangos, tal como uno haría con NumPy
df2.loc[['a','d'],["titulo", "paginas"]]
| titulo | paginas | |
|---|---|---|
| a | La piedra filosofal | 223 |
| d | El cáliz de fuego | 636 |
df2.loc[:'d',["titulo", "primer_edicion"]]
| titulo | primer_edicion | |
|---|---|---|
| a | La piedra filosofal | 1997 |
| b | La cámara secreta | 1998 |
| c | El prisionero de Azkaban | 1999 |
| d | El cáliz de fuego | 2000 |
df2.iloc[0:3, :2]
| titulo | paginas | |
|---|---|---|
| a | La piedra filosofal | 223 |
| b | La cámara secreta | 251 |
| c | El prisionero de Azkaban | 317 |
También se puede acceder a una parte de la tabla a partir de la
aplicación de una condición. Veamos por ejemplo qué libros salieron en
el siglo XXI. Para eso vemos qué elementos tienen valor de
primera_edicion mayor a 2000:
df2['primer_edicion'] > 2000
a False
b False
c False
d False
e True
f True
g True
Name: primer_edicion, dtype: bool
df2[df2['primer_edicion'] > 2000]
| titulo | paginas | primer_edicion | |
|---|---|---|---|
| e | La orden del Fénix | 766 | 2003 |
| f | El misterio del príncipe | 607 | 2005 |
| g | Las reliquias de la muerte | 607 | 2007 |
print(type(df2[df2['primer_edicion'] > 2000]))
<class 'pandas.core.frame.DataFrame'>
Modificando valores
Retomemos nuestro DataFrame inicial y agreguémosle una columna con
la calificación promedio de cada libro dada por los lectores:
df = pd.DataFrame(data)
display(df)
| Titulo | Año de edición | Páginas | |
|---|---|---|---|
| 0 | La piedra filosofal | 1997 | 223 |
| 1 | La cámara secreta | 1998 | 251 |
| 2 | El prisionero de Azkaban | 1999 | 317 |
| 3 | El cáliz de fuego | 2000 | 636 |
| 4 | La orden del Fénix | 2003 | 766 |
| 5 | El misterio del príncipe | 2005 | 607 |
| 6 | Las reliquias de la muerte | 2007 | 607 |
df['Calificación'] = [4.5, 4.3, 4.6, 4.8, 4.7, 4.9, 4.9]
df
| Titulo | Año de edición | Páginas | Calificación | |
|---|---|---|---|---|
| 0 | La piedra filosofal | 1997 | 223 | 4.5 |
| 1 | La cámara secreta | 1998 | 251 | 4.3 |
| 2 | El prisionero de Azkaban | 1999 | 317 | 4.6 |
| 3 | El cáliz de fuego | 2000 | 636 | 4.8 |
| 4 | La orden del Fénix | 2003 | 766 | 4.7 |
| 5 | El misterio del príncipe | 2005 | 607 | 4.9 |
| 6 | Las reliquias de la muerte | 2007 | 607 | 4.9 |
Supongamos que queremos agregar una columna Siglo con el siglo en
que fue escrito cada libro. Empecemos creando una columna:
df['Siglo'] = ""
df
| Titulo | Año de edición | Páginas | Calificación | Siglo | |
|---|---|---|---|---|---|
| 0 | La piedra filosofal | 1997 | 223 | 4.5 | |
| 1 | La cámara secreta | 1998 | 251 | 4.3 | |
| 2 | El prisionero de Azkaban | 1999 | 317 | 4.6 | |
| 3 | El cáliz de fuego | 2000 | 636 | 4.8 | |
| 4 | La orden del Fénix | 2003 | 766 | 4.7 | |
| 5 | El misterio del príncipe | 2005 | 607 | 4.9 | |
| 6 | Las reliquias de la muerte | 2007 | 607 | 4.9 |
Esto crea la columna Siglo que no existe en la tabla, y le asigna el
valor "" a todas las filas. Podríamos haber completado la columna
con una lista con la misma cantidad de filas que la tabla, pero mejor
vamos a seleccionar aquellos libros del siglo XX, y completaremos los
correspondientes valores en la nueva columna. Lo mismo haremos con los
del siglo XXI:
df.loc[df['Año de edición'] <= 2000, 'Siglo'] = 'XX'
df.loc[df['Año de edición'] > 2000, 'Siglo'] = 'XXI'
df
| Titulo | Año de edición | Páginas | Calificación | Siglo | |
|---|---|---|---|---|---|
| 0 | La piedra filosofal | 1997 | 223 | 4.5 | XX |
| 1 | La cámara secreta | 1998 | 251 | 4.3 | XX |
| 2 | El prisionero de Azkaban | 1999 | 317 | 4.6 | XX |
| 3 | El cáliz de fuego | 2000 | 636 | 4.8 | XX |
| 4 | La orden del Fénix | 2003 | 766 | 4.7 | XXI |
| 5 | El misterio del príncipe | 2005 | 607 | 4.9 | XXI |
| 6 | Las reliquias de la muerte | 2007 | 607 | 4.9 | XXI |
Uno podría hacer algo como lo siguiente (se llama chaining):
df.loc[df['Año de edición'] <= 2000]['Siglo'] = 'siglo XX'
df
/tmp/ipykernel_6835/1865813456.py:1: SettingWithCopyWarning:
A value is trying to be set on a copy of a slice from a DataFrame.
Try using .loc[row_indexer,col_indexer] = value instead
See the caveats in the documentation: https://pandas.pydata.org/pandas-docs/stable/user_guide/indexing.html#returning-a-view-versus-a-copy
df.loc[df['Año de edición'] <= 2000]['Siglo'] = 'siglo XX'
| Titulo | Año de edición | Páginas | Calificación | Siglo | |
|---|---|---|---|---|---|
| 0 | La piedra filosofal | 1997 | 223 | 4.5 | XX |
| 1 | La cámara secreta | 1998 | 251 | 4.3 | XX |
| 2 | El prisionero de Azkaban | 1999 | 317 | 4.6 | XX |
| 3 | El cáliz de fuego | 2000 | 636 | 4.8 | XX |
| 4 | La orden del Fénix | 2003 | 766 | 4.7 | XXI |
| 5 | El misterio del príncipe | 2005 | 607 | 4.9 | XXI |
| 6 | Las reliquias de la muerte | 2007 | 607 | 4.9 | XXI |
¿Qué pasó? Por la forma en que pandas obtiene los valores pedidos
por la máscara df['Año de edición'] <= 2000, el proceso de
chaining genera un DataFrame intermedio que es el que se pretende
modificar con ['Siglo']. El resultado es que df no se modifica.
Para evitar estos inconvenientes, no se recomienda usar chaining al hacer modificaciones en los
DataFrames.
Para borrar filas o columnas, usamos .drop:
df = df.drop(3)
df
| Titulo | Año de edición | Páginas | Calificación | Siglo | |
|---|---|---|---|---|---|
| 0 | La piedra filosofal | 1997 | 223 | 4.5 | XX |
| 1 | La cámara secreta | 1998 | 251 | 4.3 | XX |
| 2 | El prisionero de Azkaban | 1999 | 317 | 4.6 | XX |
| 4 | La orden del Fénix | 2003 | 766 | 4.7 | XXI |
| 5 | El misterio del príncipe | 2005 | 607 | 4.9 | XXI |
| 6 | Las reliquias de la muerte | 2007 | 607 | 4.9 | XXI |
df
| Titulo | Año de edición | Páginas | Calificación | Siglo | |
|---|---|---|---|---|---|
| 0 | La piedra filosofal | 1997 | 223 | 4.5 | XX |
| 1 | La cámara secreta | 1998 | 251 | 4.3 | XX |
| 2 | El prisionero de Azkaban | 1999 | 317 | 4.6 | XX |
| 4 | La orden del Fénix | 2003 | 766 | 4.7 | XXI |
| 5 | El misterio del príncipe | 2005 | 607 | 4.9 | XXI |
| 6 | Las reliquias de la muerte | 2007 | 607 | 4.9 | XXI |
df.drop('Siglo',axis=1) # no modifica el dataframe original
display(df)
df.drop('Siglo',axis=1, inplace=True) # modifica el dataframe original
display(df)
| Titulo | Año de edición | Páginas | Calificación | Siglo | |
|---|---|---|---|---|---|
| 0 | La piedra filosofal | 1997 | 223 | 4.5 | XX |
| 1 | La cámara secreta | 1998 | 251 | 4.3 | XX |
| 2 | El prisionero de Azkaban | 1999 | 317 | 4.6 | XX |
| 4 | La orden del Fénix | 2003 | 766 | 4.7 | XXI |
| 5 | El misterio del príncipe | 2005 | 607 | 4.9 | XXI |
| 6 | Las reliquias de la muerte | 2007 | 607 | 4.9 | XXI |
| Titulo | Año de edición | Páginas | Calificación | |
|---|---|---|---|---|
| 0 | La piedra filosofal | 1997 | 223 | 4.5 |
| 1 | La cámara secreta | 1998 | 251 | 4.3 |
| 2 | El prisionero de Azkaban | 1999 | 317 | 4.6 |
| 4 | La orden del Fénix | 2003 | 766 | 4.7 |
| 5 | El misterio del príncipe | 2005 | 607 | 4.9 |
| 6 | Las reliquias de la muerte | 2007 | 607 | 4.9 |
Supongamos ahora que tenemos un DataFrame (o una serie) con el
número de capítulos de los libros:
capitulos_data = {
"Titulo": [
"The Philosopher's Stone",
"The Chamber of Secrets",
"The Prisoner of Azkaban",
"The Goblet of Fire",
"The Order of the Phoenix",
"The Half-Blood Prince",
"The Deathly Hallows"
],
"Capítulos": [17, 18, 22, 37, 38, 30, 36]
}
capitulos = pd.DataFrame(capitulos_data)
display(capitulos)
| Titulo | Capítulos | |
|---|---|---|
| 0 | The Philosopher's Stone | 17 |
| 1 | The Chamber of Secrets | 18 |
| 2 | The Prisoner of Azkaban | 22 |
| 3 | The Goblet of Fire | 37 |
| 4 | The Order of the Phoenix | 38 |
| 5 | The Half-Blood Prince | 30 |
| 6 | The Deathly Hallows | 36 |
Quisiéramos agregar los datos de los capítulos a la primer tabla:
df['Capítulos'] = capitulos['Capítulos']
display(df)
| Titulo | Año de edición | Páginas | Calificación | Capítulos | |
|---|---|---|---|---|---|
| 0 | La piedra filosofal | 1997 | 223 | 4.5 | 17 |
| 1 | La cámara secreta | 1998 | 251 | 4.3 | 18 |
| 2 | El prisionero de Azkaban | 1999 | 317 | 4.6 | 22 |
| 4 | La orden del Fénix | 2003 | 766 | 4.7 | 38 |
| 5 | El misterio del príncipe | 2005 | 607 | 4.9 | 30 |
| 6 | Las reliquias de la muerte | 2007 | 607 | 4.9 | 36 |
La columna queda agregada correctamente, y no se agrega el dato en el
libro 3 porque ya no está en df. Viceversa, si queremos agregar el
número de páginas desde df a la tabla de capítulos:
capitulos['Páginas'] = df['Páginas']
display(capitulos)
| Titulo | Capítulos | Páginas | |
|---|---|---|---|
| 0 | The Philosopher's Stone | 17 | 223.0 |
| 1 | The Chamber of Secrets | 18 | 251.0 |
| 2 | The Prisoner of Azkaban | 22 | 317.0 |
| 3 | The Goblet of Fire | 37 | NaN |
| 4 | The Order of the Phoenix | 38 | 766.0 |
| 5 | The Half-Blood Prince | 30 | 607.0 |
| 6 | The Deathly Hallows | 36 | 607.0 |
Vemos como naturalmente completa con Nan el dato inexistente. Este
comportamiento es consistente en todos los casos, ya sea copiando una
sola columna o varias, o copiando partes de la tabla utilizando listas o
rangos para su definición.
Operaciones con DataFrames
Vamos a ver algunas operaciones de las muchas que se pueden hacer con
DataFrames.
Recordemos que la representación interna de los
DataFrames es equivalente a los arreglos de NumPy, con lo cual siempre se podrán realizar cálculos a través de las funciones y métodos que provee dicha biblioteca.
Aritméticas
s1 = pd.DataFrame(np.arange(1,11).reshape(5,2), index = ['a', 'b', 'c', 'd', 'e'],columns=['A', 'B'])
s2 = pd.DataFrame(np.arange(1,11).reshape(2,5).T, index = ['a', 'g', 'h', 'd', 'e'],columns=['A', 'B'])
display(s1,s2)
| A | B | |
|---|---|---|
| a | 1 | 2 |
| b | 3 | 4 |
| c | 5 | 6 |
| d | 7 | 8 |
| e | 9 | 10 |
| A | B | |
|---|---|---|
| a | 1 | 6 |
| g | 2 | 7 |
| h | 3 | 8 |
| d | 4 | 9 |
| e | 5 | 10 |
1/s1
| A | B | |
|---|---|---|
| a | 1.000000 | 0.500000 |
| b | 0.333333 | 0.250000 |
| c | 0.200000 | 0.166667 |
| d | 0.142857 | 0.125000 |
| e | 0.111111 | 0.100000 |
s1 - 42
| A | B | |
|---|---|---|
| a | -41 | -40 |
| b | -39 | -38 |
| c | -37 | -36 |
| d | -35 | -34 |
| e | -33 | -32 |
Al usar dos o más DataFrames, el proceso de alineamiento de datos
de acuerdo a las etiquetas de las filas introduce automáticamente los
datos faltantes. Por lo tanto, aquellos valores que faltan no serán, en
este caso, sumados y resultarán también en un dato inexistente:
s1+s2
| A | B | |
|---|---|---|
| a | 2.0 | 8.0 |
| b | NaN | NaN |
| c | NaN | NaN |
| d | 11.0 | 17.0 |
| e | 14.0 | 20.0 |
| g | NaN | NaN |
| h | NaN | NaN |
Es posible operar con DataFrames asignando un valor definido a los
datos inexistentes, con el argumento fill_value:
# Función de convenciencia para mostrar dos tablas una al lado de la otra
from IPython.display import display, HTML
def display_side_by_side(dfs:list, captions:list):
"""Display tables side by side to save vertical space
Input:
dfs: list of pandas.DataFrame
captions: list of table captions
"""
output = ""
combined = dict(zip(captions, dfs))
for caption, df in combined.items():
output += df.style.set_table_attributes("style='display:inline'").set_caption(caption)._repr_html_()
output += "\xa0\xa0\xa0"
display(HTML(output))
display_side_by_side([s1,s2],['s1','s2'])
| A | B | |
|---|---|---|
| a | 1 | 2 |
| b | 3 | 4 |
| c | 5 | 6 |
| d | 7 | 8 |
| e | 9 | 10 |
| A | B | |
|---|---|---|
| a | 1 | 6 |
| g | 2 | 7 |
| h | 3 | 8 |
| d | 4 | 9 |
| e | 5 | 10 |
display_side_by_side([s1,s2,s2.add(s1, fill_value=0)],['s1','s2','s2+s1'])
| A | B | |
|---|---|---|
| a | 1 | 2 |
| b | 3 | 4 |
| c | 5 | 6 |
| d | 7 | 8 |
| e | 9 | 10 |
| A | B | |
|---|---|---|
| a | 1 | 6 |
| g | 2 | 7 |
| h | 3 | 8 |
| d | 4 | 9 |
| e | 5 | 10 |
| A | B | |
|---|---|---|
| a | 2.000000 | 8.000000 |
| b | 3.000000 | 4.000000 |
| c | 5.000000 | 6.000000 |
| d | 11.000000 | 17.000000 |
| e | 14.000000 | 20.000000 |
| g | 2.000000 | 7.000000 |
| h | 3.000000 | 8.000000 |
s3 = pd.DataFrame(np.array([1,2]*5).reshape(5,2), index = ['a', 'g', 'h', 'd', 'e'],columns=['A', 'C'])
display(s3)
| A | C | |
|---|---|---|
| a | 1 | 2 |
| g | 1 | 2 |
| h | 1 | 2 |
| d | 1 | 2 |
| e | 1 | 2 |
La misma alineación de datos ocurre con las columnas:
display_side_by_side([s1,s3],['s1','s3'])
| A | B | |
|---|---|---|
| a | 1 | 2 |
| b | 3 | 4 |
| c | 5 | 6 |
| d | 7 | 8 |
| e | 9 | 10 |
| A | C | |
|---|---|---|
| a | 1 | 2 |
| g | 1 | 2 |
| h | 1 | 2 |
| d | 1 | 2 |
| e | 1 | 2 |
s1+s3
| A | B | C | |
|---|---|---|---|
| a | 2.0 | NaN | NaN |
| b | NaN | NaN | NaN |
| c | NaN | NaN | NaN |
| d | 8.0 | NaN | NaN |
| e | 10.0 | NaN | NaN |
| g | NaN | NaN | NaN |
| h | NaN | NaN | NaN |
En el caso anterior, la columna B (C) no está en el DataFrame s3
(s1), por lo tanto se completan como inexistentes. Si se utiliza
fill_value, se completan aquellos datos con fill_value que están
en alguno de los DataFrames.
Si el dato no existe en ninguno de los
DataFrames, permanece como inexistente
display_side_by_side([s1,s3,s3.add(s1, fill_value=0)],['s1','s3','s3+s1'])
| A | B | |
|---|---|---|
| a | 1 | 2 |
| b | 3 | 4 |
| c | 5 | 6 |
| d | 7 | 8 |
| e | 9 | 10 |
| A | C | |
|---|---|---|
| a | 1 | 2 |
| g | 1 | 2 |
| h | 1 | 2 |
| d | 1 | 2 |
| e | 1 | 2 |
| A | B | C | |
|---|---|---|---|
| a | 2.000000 | 2.000000 | 2.000000 |
| b | 3.000000 | 4.000000 | nan |
| c | 5.000000 | 6.000000 | nan |
| d | 8.000000 | 8.000000 | 2.000000 |
| e | 10.000000 | 10.000000 | 2.000000 |
| g | 1.000000 | nan | 2.000000 |
| h | 1.000000 | nan | 2.000000 |
s3.mul(s1,fill_value=1) # s3*s1
| A | B | C | |
|---|---|---|---|
| a | 1.0 | 2.0 | 2.0 |
| b | 3.0 | 4.0 | NaN |
| c | 5.0 | 6.0 | NaN |
| d | 7.0 | 8.0 | 2.0 |
| e | 9.0 | 10.0 | 2.0 |
| g | 1.0 | NaN | 2.0 |
| h | 1.0 | NaN | 2.0 |
En resumen, las operaciones realizan una unión entre los
DataFrames, y luego, en aquellos casos en que es factible, se
ejecuta la operación elemento a elemento.
Operaciones entre DataFrame y Series
También se pueden hacer operaciones matemáticas entre DataFrame y
Series de manera similar. Sin embargo, hay que aclarar cómo se
desarrollan las operaciones dado que mientras DataFrame representa
una estructura 2D, Series es unidimensional. Por defecto, pandas
propaga la serie por filas, al igual que NumPy. Esta extensión
automática de los datos se conoce como broadcasting:
arr2D = np.arange(12).reshape(4,3)
arr1D = np.array([3,2,1])
print('arr2D\n',arr2D)
print('arr1D\n',arr1D)
print('Broadcast')
print(np.broadcast_to(arr1D, arr2D.shape))
print(arr2D-arr1D)
arr2D
[[ 0 1 2]
[ 3 4 5]
[ 6 7 8]
[ 9 10 11]]
arr1D
[3 2 1]
Broadcast
[[3 2 1]
[3 2 1]
[3 2 1]
[3 2 1]]
[[-3 -1 1]
[ 0 2 4]
[ 3 5 7]
[ 6 8 10]]
df = pd.DataFrame(arr2D, columns = ['A', 'B', 'C'])
v = pd.Series(arr1D, index = ['A', 'B', 'C'])
display_side_by_side([df, pd.DataFrame(v),pd.DataFrame(np.broadcast_to(v, df.shape), columns=['A','B','C'])], ['df', 'v', 'broadcast'])
df - arr1D # broadcasting por filas, DataFrame - array 1D
| A | B | C | |
|---|---|---|---|
| 0 | 0 | 1 | 2 |
| 1 | 3 | 4 | 5 |
| 2 | 6 | 7 | 8 |
| 3 | 9 | 10 | 11 |
| 0 | |
|---|---|
| A | 3 |
| B | 2 |
| C | 1 |
| A | B | C | |
|---|---|---|---|
| 0 | 3 | 2 | 1 |
| 1 | 3 | 2 | 1 |
| 2 | 3 | 2 | 1 |
| 3 | 3 | 2 | 1 |
| A | B | C | |
|---|---|---|---|
| 0 | -3 | -1 | 1 |
| 1 | 0 | 2 | 4 |
| 2 | 3 | 5 | 7 |
| 3 | 6 | 8 | 10 |
df - v # broadcasting por filas, DataFrame - Serie
| A | B | C | |
|---|---|---|---|
| 0 | -3 | -1 | 1 |
| 1 | 0 | 2 | 4 |
| 2 | 3 | 5 | 7 |
| 3 | 6 | 8 | 10 |
v - df
| A | B | C | |
|---|---|---|---|
| 0 | 3 | 1 | -1 |
| 1 | 0 | -2 | -4 |
| 2 | -3 | -5 | -7 |
| 3 | -6 | -8 | -10 |
Se puede hacer el broadcasting en forma explícita, usando el argumento
axis: - Por columnas: axis = "index" (o axis = 0) - Por
filas: axis = "columns" (o axis = 1) que indica qué índice debe
alinearse.
w = df['A']
display_side_by_side([df, pd.DataFrame(w)], ['df', 'w'])
df.sub(w, axis='index') # alinea por filas, broadcasting por columnas, DataFrame - Serie
| A | B | C | |
|---|---|---|---|
| 0 | 0 | 1 | 2 |
| 1 | 3 | 4 | 5 |
| 2 | 6 | 7 | 8 |
| 3 | 9 | 10 | 11 |
| A | |
|---|---|
| 0 | 0 |
| 1 | 3 |
| 2 | 6 |
| 3 | 9 |
| A | B | C | |
|---|---|---|---|
| 0 | 0 | 1 | 2 |
| 1 | 0 | 1 | 2 |
| 2 | 0 | 1 | 2 |
| 3 | 0 | 1 | 2 |
df.sub(w, axis=0) # alinea por filas, broadcasting por columnas, DataFrame - Serie, idem axis='index'
| A | B | C | |
|---|---|---|---|
| 0 | 0 | 1 | 2 |
| 1 | 0 | 1 | 2 |
| 2 | 0 | 1 | 2 |
| 3 | 0 | 1 | 2 |
t = pd.Series([3,2,1,0], index = ['A', 'B', 'C', 'D'])
display_side_by_side([df, pd.DataFrame(t)], ['df', 't'])
| A | B | C | |
|---|---|---|---|
| 0 | 0 | 1 | 2 |
| 1 | 3 | 4 | 5 |
| 2 | 6 | 7 | 8 |
| 3 | 9 | 10 | 11 |
| 0 | |
|---|---|
| A | 3 |
| B | 2 |
| C | 1 |
| D | 0 |
df.sub(t, axis=1) # alínea por columnas, broadcasting por filas explícito, DataFrame - Serie
| A | B | C | D | |
|---|---|---|---|---|
| 0 | -3 | -1 | 1 | NaN |
| 1 | 0 | 2 | 4 | NaN |
| 2 | 3 | 5 | 7 | NaN |
| 3 | 6 | 8 | 10 | NaN |
En este último caso, dado que los índices de w y df son todos
distintos, se realiza la unión y se completa con NaN.
En Resumen: el eje (
axis) es aquel en el cual se van a alinear los índices. Así,axis=0implica emparejar los índices de filas y propagar por columnas. De la misma manera,axis=1implica emparejar los índices de columnas y propagar por filas.
Orden
Para ordenar DataFrames se usa el método .sort_index()
dfo = pd.DataFrame(np.random.rand(4,3), index= ['c','d','a','b'], columns = ['B', 'C', 'A'])
dfo
| B | C | A | |
|---|---|---|---|
| c | 0.708606 | 0.269161 | 0.674871 |
| d | 0.760163 | 0.806327 | 0.364229 |
| a | 0.977312 | 0.865779 | 0.916780 |
| b | 0.167906 | 0.519749 | 0.732298 |
dfo.sort_index() # ordena por los índices de filas
| B | C | A | |
|---|---|---|---|
| a | 0.977312 | 0.865779 | 0.916780 |
| b | 0.167906 | 0.519749 | 0.732298 |
| c | 0.708606 | 0.269161 | 0.674871 |
| d | 0.760163 | 0.806327 | 0.364229 |
dfo.sort_index(axis=1) # ordena por los índices de columnas
| A | B | C | |
|---|---|---|---|
| c | 0.674871 | 0.708606 | 0.269161 |
| d | 0.364229 | 0.760163 | 0.806327 |
| a | 0.916780 | 0.977312 | 0.865779 |
| b | 0.732298 | 0.167906 | 0.519749 |
Pasando el argumento ascending (booleano) se puede cambiar el orden
por defecto, que corresponde a ascending=True. Se puede ordenar
también de acuerdo a los valores de una (o múltiples) columnas
utilizando el método sort_values:
dfo.sort_values('B')
| B | C | A | |
|---|---|---|---|
| b | 0.167906 | 0.519749 | 0.732298 |
| c | 0.708606 | 0.269161 | 0.674871 |
| d | 0.760163 | 0.806327 | 0.364229 |
| a | 0.977312 | 0.865779 | 0.916780 |
En el caso en que existieran valores faltantes, estos se ordenan al
final por defecto, excepto que se pase el argumento opcional
na_position='first'.
dfo.loc['c','A'] = None
dfo.loc['a','C'] = None
dfo
| B | C | A | |
|---|---|---|---|
| c | 0.708606 | 0.269161 | NaN |
| d | 0.760163 | 0.806327 | 0.364229 |
| a | 0.977312 | NaN | 0.916780 |
| b | 0.167906 | 0.519749 | 0.732298 |
dfo.sort_values('C')
| B | C | A | |
|---|---|---|---|
| c | 0.708606 | 0.269161 | NaN |
| b | 0.167906 | 0.519749 | 0.732298 |
| d | 0.760163 | 0.806327 | 0.364229 |
| a | 0.977312 | NaN | 0.916780 |
dfo.sort_values('C',na_position='first') # coloca los NaN al principio
| B | C | A | |
|---|---|---|---|
| a | 0.977312 | NaN | 0.916780 |
| c | 0.708606 | 0.269161 | NaN |
| b | 0.167906 | 0.519749 | 0.732298 |
| d | 0.760163 | 0.806327 | 0.364229 |
Una operación útil es ranking, esto es, asignar un valor desde 1 hasta el número de datos de acuerdo a un orden, desde el valor más bajo al más alto:
s = pd.Series(np.random.rand(6))
s
0 0.395530
1 0.030781
2 0.572166
3 0.195011
4 0.986553
5 0.178980
dtype: float64
s.rank()
0 4.0
1 1.0
2 5.0
3 3.0
4 6.0
5 2.0
dtype: float64
display_side_by_side([dfo, df.rank(axis=0)],['df','df.rank->0']) # ordena a lo largo de columnas
display_side_by_side([dfo, df.rank(axis=1)],['df','df.rank->1']) # ordena a lo largo de filas
| B | C | A | |
|---|---|---|---|
| c | 0.708606 | 0.269161 | nan |
| d | 0.760163 | 0.806327 | 0.364229 |
| a | 0.977312 | nan | 0.916780 |
| b | 0.167906 | 0.519749 | 0.732298 |
| A | B | C | |
|---|---|---|---|
| 0 | 1.000000 | 1.000000 | 1.000000 |
| 1 | 2.000000 | 2.000000 | 2.000000 |
| 2 | 3.000000 | 3.000000 | 3.000000 |
| 3 | 4.000000 | 4.000000 | 4.000000 |
| B | C | A | |
|---|---|---|---|
| c | 0.708606 | 0.269161 | nan |
| d | 0.760163 | 0.806327 | 0.364229 |
| a | 0.977312 | nan | 0.916780 |
| b | 0.167906 | 0.519749 | 0.732298 |
| A | B | C | |
|---|---|---|---|
| 0 | 1.000000 | 2.000000 | 3.000000 |
| 1 | 1.000000 | 2.000000 | 3.000000 |
| 2 | 1.000000 | 2.000000 | 3.000000 |
| 3 | 1.000000 | 2.000000 | 3.000000 |
Estadística
Es muy común en el análisis de datos tener que calcular valores
estadísticos que caracterizan la muestra. pandas provee una serie de
métodos para realizar estos cálculos.
df = pd.DataFrame(np.random.rand(4,3), index= ['a','b','c','d'], columns = ['A', 'B', 'C'])
df
| A | B | C | |
|---|---|---|---|
| a | 0.020433 | 0.217049 | 0.949068 |
| b | 0.317689 | 0.684951 | 0.017181 |
| c | 0.622041 | 0.421691 | 0.776658 |
| d | 0.317763 | 0.377677 | 0.231512 |
df.describe()
| A | B | C | |
|---|---|---|---|
| count | 4.000000 | 4.000000 | 4.000000 |
| mean | 0.319482 | 0.425342 | 0.493605 |
| std | 0.245614 | 0.194137 | 0.440922 |
| min | 0.020433 | 0.217049 | 0.017181 |
| 25% | 0.243375 | 0.337520 | 0.177930 |
| 50% | 0.317726 | 0.399684 | 0.504085 |
| 75% | 0.393833 | 0.487506 | 0.819760 |
| max | 0.622041 | 0.684951 | 0.949068 |
Cada una de las magnitudes se corresponde a algún parámetro estadístico.
Estas operaciones devuelven un nuevo DataFrame. Por ejemplo:
df.sum()
A 1.277927
B 1.701367
C 1.974419
dtype: float64
retorna un DataFrame donde los índices corresponden a las etiquetas
de cada columna.
Por defecto, los valores inexistentes (NA, Null, etc.) no son tenidos en cuenta al realizar estas operaciones.
df['A'].sum()
1.2779272965340271
Aplicación de funciones y map
Es posible también aplicar funciones, por ejemplo
dff = pd.DataFrame(np.random.rand(4,3), index= ['a','b','c','d'], columns = ['A', 'B', 'C'])
dff
| A | B | C | |
|---|---|---|---|
| a | 0.397259 | 0.852559 | 0.395104 |
| b | 0.546917 | 0.361448 | 0.622568 |
| c | 0.425802 | 0.731688 | 0.053037 |
| d | 0.761319 | 0.653025 | 0.183615 |
def f(x):
return x.max() - x.min()
dff.apply(f) # aplica la función a cada columna, por defecto axis=0
A 0.364060
B 0.491111
C 0.569531
dtype: float64
dff.apply(f, axis=1) # aplica la función a cada fila, axis=1
a 0.457455
b 0.261119
c 0.678651
d 0.577704
dtype: float64
dff.apply(lambda x: x.max()-x.min(), axis=1) # por columnas, igual que df.apply(f, axis='columns')
a 0.457455
b 0.261119
c 0.678651
d 0.577704
dtype: float64
La siguiente operación calcular el promedio de las columnas de un
DataFrame e incorporarlos como una columna nueva:
dff['mean'] = dff.mean(axis=1)
dff
| A | B | C | mean | |
|---|---|---|---|---|
| a | 0.397259 | 0.852559 | 0.395104 | 0.548308 |
| b | 0.546917 | 0.361448 | 0.622568 | 0.510311 |
| c | 0.425802 | 0.731688 | 0.053037 | 0.403509 |
| d | 0.761319 | 0.653025 | 0.183615 | 0.532653 |
Cuál es el resultado de dff['mean'] = dff.mean(axis=0)?
dff.drop('mean', axis=1, inplace=True) # elimina la columna 'mean'
dff.mean(axis=0)
A 0.532824
B 0.649680
C 0.313581
dtype: float64
dff['mean'] = dff.mean(axis=0)
dff
| A | B | C | mean | |
|---|---|---|---|---|
| a | 0.397259 | 0.852559 | 0.395104 | NaN |
| b | 0.546917 | 0.361448 | 0.622568 | NaN |
| c | 0.425802 | 0.731688 | 0.053037 | NaN |
| d | 0.761319 | 0.653025 | 0.183615 | NaN |
Vemos que, como no existen los índices A, B, C, que
corresponden a las columnas que acabamos de calcular, asigna NaN en
los índices a, b, c, d
Puede ser necesario aplicar determinada función a todos los valores del
DataFrame, con map:
def round2(x):
return round(x, 2)
display_side_by_side([df, df.map(round2)], ['df', 'df.map(round2)']) # aplica la función a cada elemento del DataFrame
| A | B | C | |
|---|---|---|---|
| a | 0.020433 | 0.217049 | 0.949068 |
| b | 0.317689 | 0.684951 | 0.017181 |
| c | 0.622041 | 0.421691 | 0.776658 |
| d | 0.317763 | 0.377677 | 0.231512 |
| A | B | C | |
|---|---|---|---|
| a | 0.020000 | 0.220000 | 0.950000 |
| b | 0.320000 | 0.680000 | 0.020000 |
| c | 0.620000 | 0.420000 | 0.780000 |
| d | 0.320000 | 0.380000 | 0.230000 |
Ejercicios 14 (c)
El archivo ‘clima argentina 1981 2010.txt’ contiene datos climáticos significativos de las distintas estaciones meteorológicas del Servicio Meteorológico Nacional.
Inspeccione el archivo y diseñe un tipo
DataFrameadecuado para contener dichos datos.Lea el archivo y cree el
DataFrameque diseño en el item anterior.Cree una función para obtener el promedio anual de las magnitudes climáticas referidas en el archivo. La función debe recibir como argumentos el
DataFrame, el nombre de la estación meteorológica y la magnitud, y devolver un valor (float) con el promedio.Genere un
DataFrameque represente todas las magnitudes promedio para cada estación meteorológica, usando la función del item anterior.Cree funciones para poder realizar gráficos comparativos de los datos meteorológicos. En particular, reproduzca
El gráfico de Valores Medios de Temperatura y Precipitación
El gráfico de Valores extremos de Temperatura, usando la temperatura máxima y la mínima de cada mes.
que se pueden ver en el sitio de estadísticas del Servicio Meteorológico Nacional.